
- May -akda Miles Stephen stephen@answers-science.com.
- Public 2023-12-15 23:41.
- Huling binago 2025-01-22 17:11.
Ito ay isang negatibo paglamlam pamamaraan na mahalagang ginagamit upang makilala ang pagkakaroon ng mga kapsula . Dahil sa pagiging acidic nito, ang tinta ng India (o Congo pula , nigrosin) mga mantsa madilim ang background. Sa kabilang banda, ang crystal violet ay ginagamit para sa maraming mga kadahilanan kabilang ang: Upang kumilos bilang isang fixative. Dagdagan ang lakas ng pagtagos.
Katulad nito, ano ang layunin ng paglamlam ng kapsula?
Pangunahing layunin ng capsule stain ay upang makilala capsular materyal mula sa bacterial cell. A kapsula ay isang gelatinous na panlabas na layer na itinago ng bacterial cell at pumapalibot at dumidikit sa cell wall. Karamihan mga kapsula ay binubuo ng polysaccharides, ngunit ang ilan ay binubuo ng polypeptides.
Gayundin, maaari mo bang gamitin ang Congo red para sa positibong paglamlam? Congo pula hindi maaaring gamitin para sa direkta paglamlam dahil ito ay acid mantsa (negatively charged) at ang layunin ng isang direktang mantsa ay gamitin isang basic mga mantsa ( positibo sinisingil) upang mantsa ang bacterium mismo dahil sa isang negatibong singil at positibo singil ay naaakit sa bawat isa, ang negatibo at negatibong singil ay nagtataboy sa isa't isa
Dito, anong mga mantsa ang ginagamit para sa paglamlam ng kapsula?
Bakterya mga kapsula ay hindi ionic, kaya hindi acidic o basic mga mantsa ay susunod sa kanilang mga ibabaw. Samakatuwid, ang pinakamahusay na paraan upang mailarawan ang mga ito ay mantsa ang background gamit ang isang acidic mantsa at sa mantsa ang cell mismo gamit ang isang basic mantsa . Gumagamit kami ng India ink at Gram crystal violet.
Paano tayo tinutulungan ng negatibong paglamlam na makita ang mga kapsula?
Bilang kahalili, positibo at negatibong paglamlam maaaring pagsamahin ang mga teknik sa tingnan ang mga kapsula : Ang positive mantsa kulay ang katawan ng cell, at ang negatibong mantsa kulay ang background ngunit hindi ang kapsula , nag-iiwan ng halo sa paligid ng bawat cell.
Inirerekumendang:
Ano ang pagkakaiba sa pagitan ng isang mapa ng pangkalahatang layunin at isang mapa ng espesyal na layunin?

Ang diin sa mga mapa ng pangkalahatang layunin ay sa lokasyon. Ang mga mapa ng pader, karamihan sa mga mapa na matatagpuan sa mga atlas, at mga mapa ng kalsada ay nasa kategoryang ito. Ang mga pampakay na mapa, na tinutukoy din bilang mga mapa na may espesyal na layunin, ay naglalarawan ng heograpikal na pamamahagi ng isang partikular na tema o phenomenon
Ano ang layunin ng heat fixation kung ano ang nangyayari kapag sobrang init ang inilapat?

Pinapatay ng heat fixation ang bacterial cells at nagiging sanhi ng pagdidikit sa mga ito sa salamin upang hindi mabanlaw. Inheat-fixing ano ang mangyayari kung sobrang init ang inilapat? Masisira nito ang istruktura ng cell
Bakit natin inaasahan na mabahiran ng kulay rosas na pula ang Gram negative bacteria sa panahon ng Gram staining procedure?

Samantalang ang gram-positive bacteria ay nabahiran ng violet bilang resulta ng pagkakaroon ng makapal na peptidoglycan layer sa mga dingding ng kanilang cell, ang gram-negative bacteria ay nabahiran ng pula, dahil sa mas manipis na peptidoglycan layer sa kanilang cell wall (isang mas makapal na peptidoglycan layer ay nagbibigay-daan para sa pagpapanatili ng mantsa, ngunit isang mas manipis na layer
Paano mo nakikilala ang Gram staining bacteria?
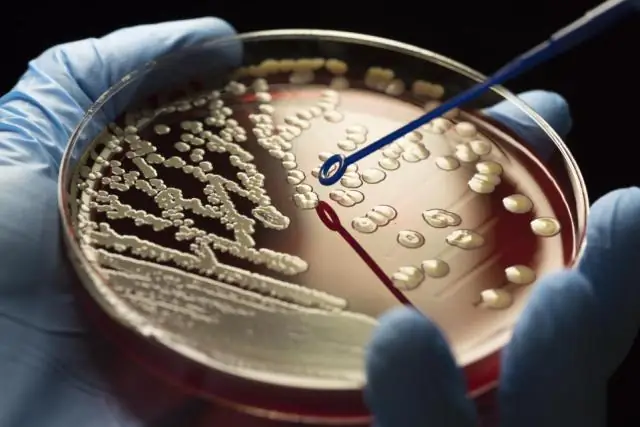
Pagkilala sa Gram Stain Ilapat ang unang mantsa (isang purple stain na tinatawag na crystal violet) sa isang heat-fixed smear ng bacterial culture. Maglagay ng yodo sa ibabaw ng crystal violet. Hugasan ang mga cell na may alkohol o acetone. Pahiran muli ang mga cell (counterstain) ng pulang pangkulay, alinman sa safranin red o basic fuchsin
Ano ang junk DNA at ano ang layunin nito?

Sa genetics, ang terminong junk DNA ay tumutukoy sa mga rehiyon ng DNA na hindi coding. Ang ilan sa noncoding DNA na ito ay ginagamit upang makagawa ng mga noncoding na bahagi ng RNA tulad ng transfer RNA, regulatory RNA at ribosomal RNA
